作者单位
摘要
The precise segmentation of different brain regions and tissues is usually a prerequisite for the detection and diagnosis of various neurological disorders in neuroscience. Considering the abundance of functional and structural dual-modality information for positron emission tomography/magnetic resonance (PET/MR) images, we propose a novel 3D whole-brain segmentation network with a cross-fusion mechanism introduced to obtain 45 brain regions. Specifically, the network processes PET and MR images simultaneously, employing UX-Net and a cross-fusion block for feature extraction and fusion in the encoder. We test our method by comparing it with other deep learning-based methods, including 3DUXNET, Swin-UNETR, UNETR, nnFormer, UNet3D, NestedUNet, ResUNet, and VNet. The experimental results demonstrate that the proposed method achieves better segmentation performance in terms of both visual and quantitative evaluation metrics and achieves more precise segmentation in three views while preserving fine details. In particular, the proposed method achieves superior quantitative results, with a Dice coefficient of 85.73% ± 0.01%, a Jaccard index of 76.68% ± 0.02%, a sensitivity of 85.00% ± 0.01%, a precision of 83.26% ± 0.03% and a Hausdorff distance (HD) of 4.4885 ± 14.85%. Moreover, the distribution and correlation of the SUV in the volume of interest (VOI) are also evaluated (PCC > 0.9), indicating consistency with the ground truth and the superiority of the proposed method. In future work, we will utilize our whole-brain segmentation method in clinical practice to assist doctors in accurately diagnosing and treating brain diseases.
翻译
不同脑区和组织的精确分割通常是神经科学中检测和诊断各种神经系统疾病的前提。考虑到正电子发射断层扫描/磁共振(PET/MR)图像的功能和结构双模态信息的丰富性,我们提出了一种新颖的三维全脑分割网络,通过引入交叉融合机制来获得45个脑区。具体而言,该网络同时处理PET和MR图像,在编码器中采用UX-Net和交叉融合块进行特征提取和融合。我们通过与其他基于深度学习的方法进行比较来测试我们的方法,包括3DUXNET、Swin-UNETR、UNETR、nnFormer、UNet3D、NestedUNet、ResUNet和VNet。实验结果表明,所提出的方法在视觉和定量评估指标方面实现了更好的分割性能,并在三个视图中实现了更精确的分割,同时保留了细节。特别是,所提出的方法在量化结果方面表现优异,Dice系数为85.73% ± 0.01%,Jaccard指数为76.68% ± 0.02%,敏感性为85.00% ± 0.01%,精确度为83.26% ± 0.03%,Hausdorff距离(HD)为4.4885 ± 14.85%。此外,还评估了感兴趣区域(VOI)中SUV的分布和相关性(PCC > 0.9),表明与真实值的一致性以及所提出方法的优越性。在未来的工作中,我们将利用我们的全脑分割方法在临床实践中帮助医生准确诊断和治疗脑疾病。
研究背景
本文聚焦于脑区分割研究,旨在解决现有方法的局限,其研究背景如下:
- 脑区分割的重要性:精准的脑区分割对神经科学研究和临床诊断意义重大,不同脑区的体积、表面积和形态与帕金森病、阿尔茨海默病等多种神经系统疾病相关。
- PET/MR成像系统的优势:正电子发射断层扫描/磁共振(PET/MR)集成成像系统结合了PET代谢成像和MR高分辨率成像的优点,是诊断脑部疾病的有效工具。
- 现有脑区分割方法的不足:手动分割脑医学图像耗时费力,且结果易受个体差异和主观因素影响;传统自动分割方法依赖手动特征工程,对图像质量和噪声敏感,对解剖变异的鲁棒性差;现有的深度学习方法多基于单模态医学图像分割,部分融合双模态的方法分割的脑区较少,融合方式简单,缺乏深度和全面的整合。 因此,本文提出一种基于交叉融合机制的自动脑分割方法,充分利用PET和MR医学图像,结合两者的功能和结构信息,以实现更精确、全面的脑区自动分割。
研究现状
- 传统方法:可分为阈值和统计方法、图像处理和数学模型方法、基于图谱的方法,但依赖手动特征工程,对图像质量和噪声敏感,对解剖变异鲁棒性差。
- 深度学习方法:基于高分辨率MR图像或代谢成像与低分辨率PET的单模态分割方法较多,部分方法开始融合PET和MR双模态信息,但存在分割脑区少、融合方法简单的问题。
提出的模型
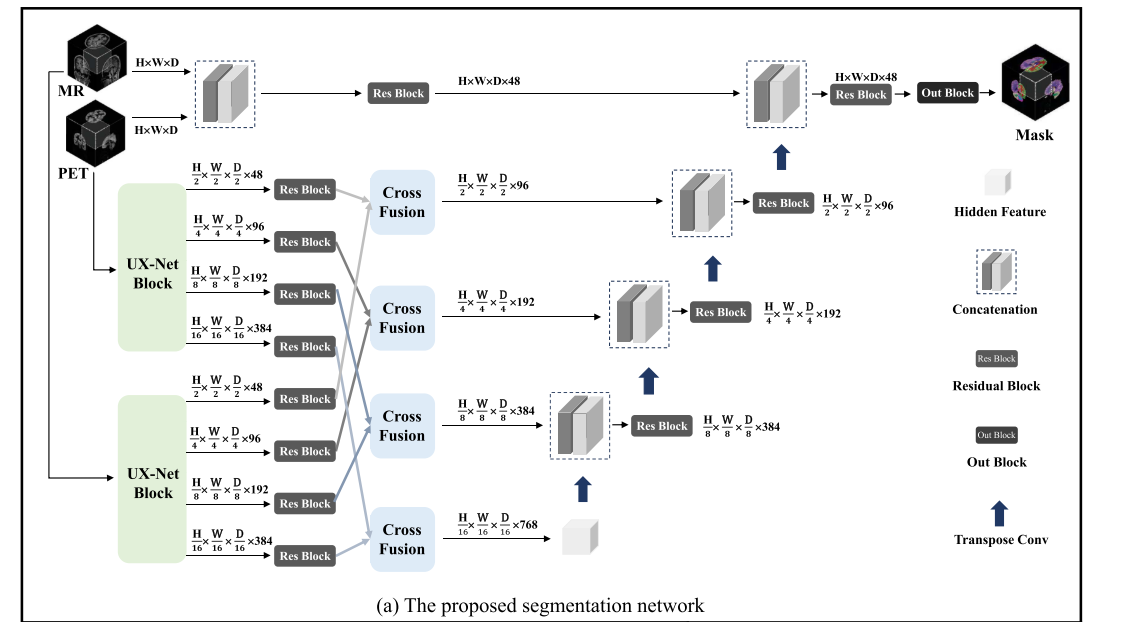
网络整体结构
模型采用经典的U形分割结构,由编码器(encoder)和解码器(decoder)组成。PET和MR图像同时作为输入,通过编码器中的特征提取模块(UX - Net block)和融合模块(cross - fusion block)进行特征提取与融合,最后由解码器完成脑分割任务。
编码器部分
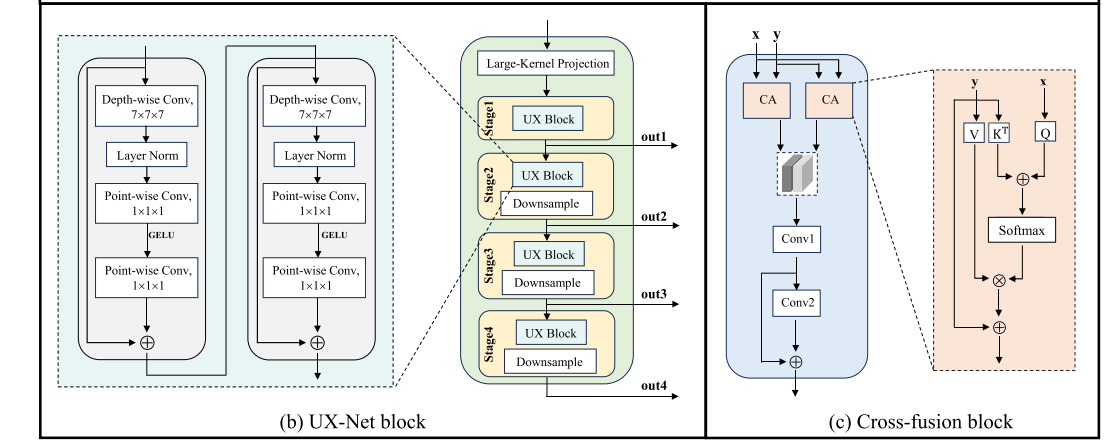
- UX-Net Block:作为特征提取器,是网络的骨干部分。该模块采用分层变压器(hierarchical transformer)的思想,使用大规模深度卷积(large-scale depthwise convolution)在不增加计算成本的情况下获取局部信息,适合分层变压器结构并保留了卷积神经网络(CNN)的归纳偏置等优点。
- 首先通过大卷积核的投影层提取图像块特征。
- 分四个阶段提取不同层次的特征,每个阶段包含两个7×7×7的大规模深度卷积和两个1×1×1的逐点卷积,以丰富特征表示并减少通道冗余。
- 每个阶段使用2×2×2的标准卷积模块将特征分辨率降低1/2,并应用层归一化(layer normalization)和高斯误差线性单元(GELU)作为激活函数。
- 最后通过包含两个批量归一化的3×3×3卷积层的残差块稳定提取的特征。
- Cross - Fusion Block:主要用于融合经过残差块处理后的PET和MR图像在四个阶段提取的特征,由两个交叉注意力块(cross - attention blocks)组成。
- 利用多通道交叉注意力机制(multichannel cross - attention mechanism)在两个不同来源的特征之间建立复杂关联,实现全面的特征交叉融合。
- 具体过程是先计算查询(query)、键(key)和值(value),得到两个特征的注意力分布,实现全局关联,再通过卷积层进一步处理输出特征。
- 通过两次交叉注意力计算(CA1和CA2)并拼接结果,最后通过残差连接得到最终的交叉融合结果。
解码器部分
编码器各阶段生成的多尺度输出通过跳跃连接(skip connections)连接到解码器,形成U形网络用于下游分割任务。
- 解码器将各阶段特征上采样并与前一阶段的特征拼接,经过残差块处理。
- 网络的原始输入和编码器的输出按从上到下顺序设置为[e0, e1, e2, e3, e4],解码器的输出按从上到下顺序设置为[d1, d2, d3, d4]。
- 以第3个解码器层为例,先对第4个解码器层的输出进行上采样,然后与编码器第2层的输出拼接,经过残差块得到该层输出。
- 最后将解码器的输出输入到包含1×1×1卷积层和softmax激活函数的残差块中,预测分割概率。
损失函数
结合了Dice系数损失(Dice coefficient loss)和交叉熵损失(cross - entropy loss)构建混合损失函数,以充分利用两种损失函数的优点,增强模型在图像分割任务中的性能。
- Dice系数损失用于衡量两个样本之间的相似度,常用于图像分割任务。
- 交叉熵损失是图像分割中广泛使用的损失函数。
- 总损失L是Dice系数损失Ldice和交叉熵损失Lce的加权和,权重为$ω$。
实验(Compared with SOTA)
数据集:使用了110名受试者的18F - FDG PET/MR脑图像,所有数据在3.0T的PET/MRI集成扫描仪上采集。利用FreeSurfer工具获取45个脑区作为真实标签(GT),并对PET和MR图像进行配准和独热编码处理。
- 训练实现
- 数据预处理:对输入图像的像素值进行最小 - 最大归一化,并裁剪前景以消除背景。同时,引入正负样本的随机裁剪,获取96×96×96的图像块。
- 数据增强:采用随机缩放、随机翻转和随机强度调整等技术增加数据多样性,缓解过拟合问题。
- 训练设置:在PyTorch框架、Windows 10系统和NVIDIA GeForce RTX 3090 24GB GPU上进行训练。训练250个轮次,批次大小设为1,学习率为1e - 4,使用AdamW动态调整训练损失,每次训练迭代输入2个96×96×96的图像块,损失权重ω设为1。
- 对比方法:将提出的方法与四种基于Transformer的模型(3DUXNET、SwinUNETR、UNETR、nnFormer)和四种基于CNN的模型(UNet3D、NestedUNet、ResUNet、VNet)进行对比。
- 评估指标:采用五种评估指标,包括Dice相似度、Jaccard系数、精确率、灵敏度和Hausdorff距离(HD),全面评估分割性能
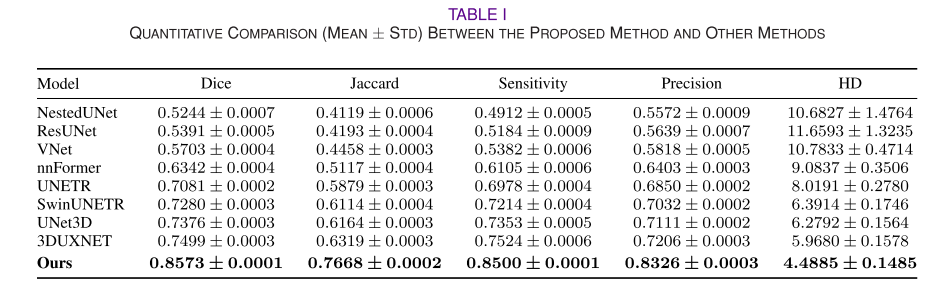
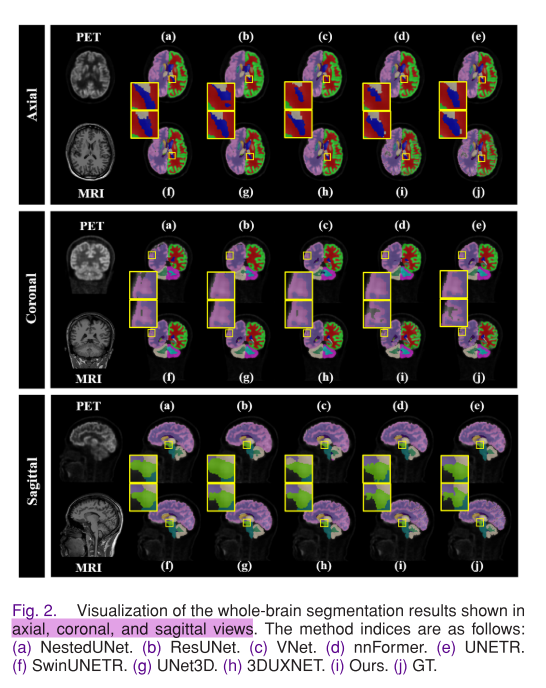
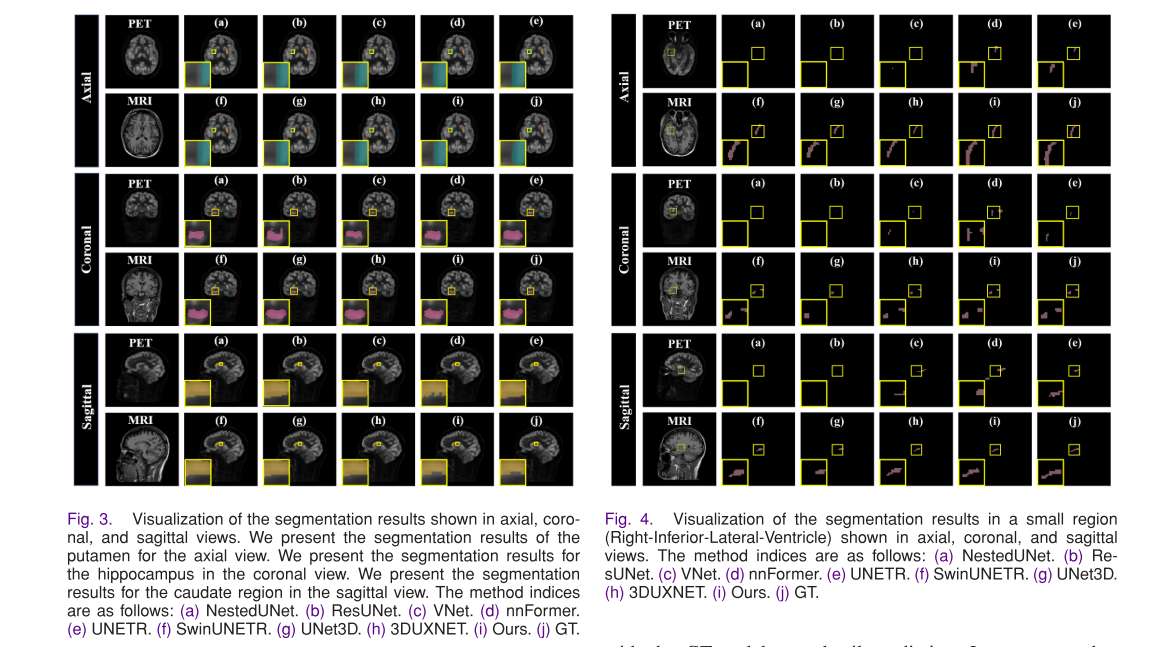
- 整体定量评估:可视化全脑分割结果,提出的方法在三个视图上的分割结果更接近GT,能完整分割45个脑区并保留细节。定量结果显示,该方法在各项指标上表现优越,相比最佳对比方法有显著提升。
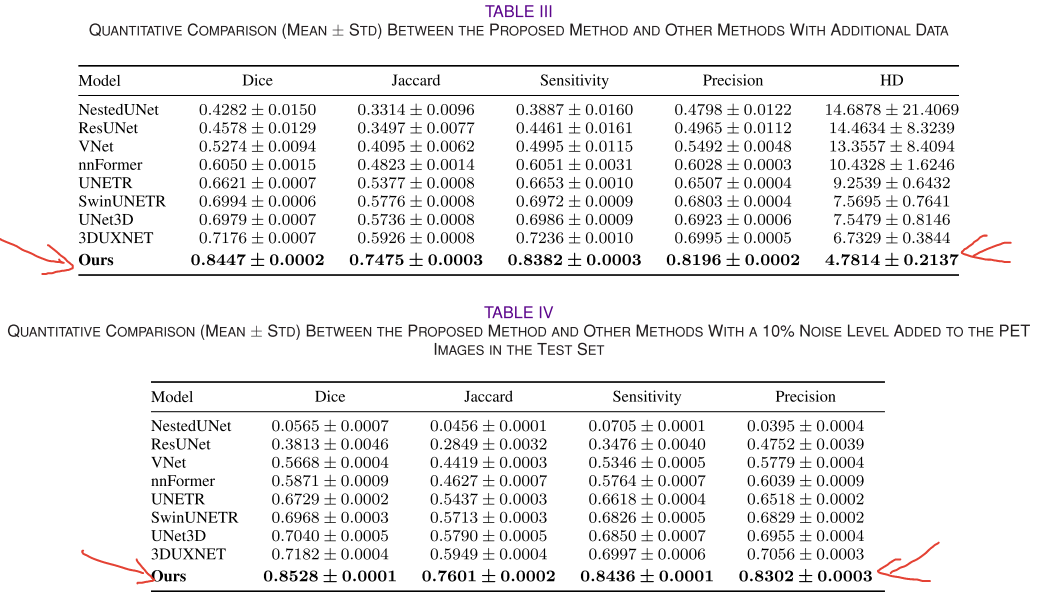
- 特定脑区评估:分析特定脑区(如壳核、海马体、尾状核和右 inferior - 侧脑室)的分割结果,提出的方法能更好地保留边缘细节,与GT的一致性更高。
- 一致性和相关性分析:分析PET图像分割结果在不同感兴趣体积(VOI)区域的标准摄取值(SUV)分布和相关性,提出的方法的SUV分布与GT最一致,相关系数PCC较高。
- 临床定量质量评估:计算肿瘤与背景比和临床容忍率,提出的方法临床容忍率最低,分割性能显著优于其他方法。
- 额外数据评估和验证
- 额外数据测试:引入40例PET和MR数据进行测试,提出的方法在额外数据集上仍保持优越性能,证明了模型的鲁棒性和通用性。
- 噪声数据测试:向测试集的PET图像中添加不同水平的噪声,提出的方法在噪声环境下仍能准确分割脑区,表现出较强的鲁棒性,而NestedUNet受噪声影响最大。
实验(Ablation Experiments)
- 消融研究和融合模块分析
- 消融分析:验证了UX - Net块和交叉融合块在模型分割效果中的关键作用,替换这两个模块会导致定量指标下降。
- 交叉融合块有效性分析:交叉融合机制在Dice分数、Jaccard系数和精确率方面取得最佳定量结果,分割性能出色。
结论
作者提出一种基于交叉融合机制的自动脑分割方法,将PET和MR多模态信息融合用于精确脑分割。实验在PET/MR数据集上开展,并与8种深度学习方法对比,从视觉、定量和临床三方面评估分割结果,证明该方法的优越性。具体而言,此方法取得了优异的定量结果,Dice得分85.73% ± 0.01%,Jaccard指数76.68% ± 0.02%等,SUV相关性评估也显示其优于其他方法,临床误差容忍率不超5%。该方法实现了精确的全脑分割,对脑部疾病的临床诊断和分析有益。未来,作者将探索该方法在其他模态图像处理任务中的应用。



