深圳大学
摘要
In the context of contemporary artificial intelligence, increasing deep learning (DL) based segmentation methods have been recently proposed for brain tumor segmentation (BraTS) via analysis of multi-modal MRI. However, known DL-based works usually directly fuse the information of different modalities at multiple stages without considering the gap between modalities, leaving much room for performance improvement. In this paper, we introduce a novel deep neural network, termed ACFNet, for accurately segmenting brain tumor in multi-modal MRI. Specifically, ACFNet has a parallel structure with three encoder-decoder streams. The upper and lower streams generate coarse predictions from individual modality, while the middle stream integrates the complementary knowledge of different modalities and bridges the gap between them to yield fine prediction. To effectively integrate the complementary information, we propose an adaptive cross-feature fusion (ACF) module at the encoder that first explores the correlation information between the feature representations from upper and lower streams and then refines the fused correlation information. To bridge the gap between the information frommulti-modal data, we propose a prediction inconsistency guidance (PIG) module at the decoder that helps the network focus more on error-prone regions through a guidance strategy when incorporating the features from the encoder. The guidance is obtained by calculating the prediction inconsistency between upper and lower streams and highlights the gap between multi-modal data. Extensive experiments on the BraTS 2020 dataset show that ACFNet is competent for the BraTS task with promising results and outperforms six mainstream competing methods.
翻译
在当代人工智能背景下,近年来提出了越来越多基于深度学习(DL)的分割方法,用于通过分析多模态MRI进行脑肿瘤分割(BraTS)。然而,已知的DL方法通常在多个阶段直接融合不同模态的信息,而没有考虑模态之间的差距,留下了很大的性能提升空间。本文中,我们介绍了一种新颖的深度神经网络,称为ACFNet,用于准确分割多模态MRI中的脑肿瘤。具体来说,ACFNet具有三条编码器-解码器流的并行结构。上下流从单个模态产生粗略预测,而中间流整合不同模态的互补知识并弥合它们之间的差距,以产生精细预测。为了有效整合互补信息,我们在编码器中提出了一种自适应交叉特征融合(ACF)模块,该模块首先探索来自上下流的特征表示之间的相关信息,然后优化融合的相关信息。为弥合多模态数据的信息差距,我们在解码器中提出了一种预测不一致性指导(PIG)模块,通过引导策略帮助网络在整合来自编码器的特征时更加关注易错区域。该指导通过计算上下流之间的预测不一致性获得,并突出多模态数据之间的差距。对BraTS 2020数据集的大量实验表明,ACFNet在BraTS任务中表现出色,取得了令人满意的结果,并超过了六种主流竞争方法。
研究背景
脑肿瘤严重威胁患者生命,其智能监测与诊断水平有待提高。磁共振成像(MRI)是脑肿瘤监测与诊断的常用手段,准确的脑肿瘤分割有助于疾病诊断和治疗方案制定,但人工标注主观、繁琐且负担重,因此设计自动化分割方法十分必要。
近年来,深度学习助力医学图像分析取得显著进展,为脑肿瘤分割提供了大量灵感。然而,脑肿瘤MRI包含多种模态,如FLAIR、T2、T1和T1ce,如何有效挖掘这些模态的信息是设计多模态脑肿瘤分割方法的关键。现有方法通常采用编码器 - 解码器结构,但大多直接融合不同模态信息,忽略了模态间的差异,即“模态差距”,这会导致各模态细节孤立,影响模型性能,也阻碍了准确描绘肿瘤边界的能力。
为解决这些问题,本文提出了自适应交叉特征融合网络(ACFNet),旨在自适应地整合和细化不同模态的特征,解决模态差距问题,实现更准确的多模态脑肿瘤分割。
研究现状
- 多模态学习:多模态数据能提供互补信息,简单拼接多模态图像输入DNN效果不佳,近期更多研究关注基于DNN或病变特征融合多模态数据。
- 医学图像分割:CNN 方法在医学图像分割中占重要地位,U-Net及其变体应用广泛,Vision Transformers也展现出良好性能,部分方法结合二者优势。
- 脑肿瘤分割:早期采用生成概率模型融合多模态信息,目前主要是2D和3D CNN方法。多模态脑肿瘤分割方法分为早期融合、晚期融合和多层融合三类,多层融合效果更好。
提出的模型
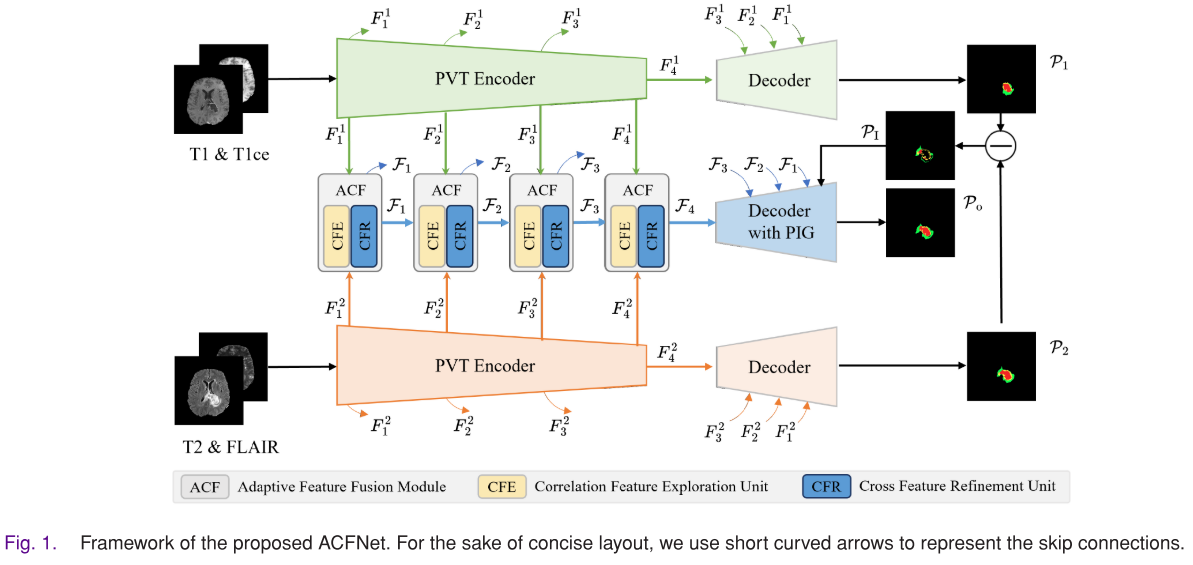
本文提出了一种名为自适应跨特征融合网络(Adaptive Cross-feature Fusion Network,ACFNet)的新型深度神经网络,用于多模态脑肿瘤分割
- 整体架构:ACFNet具有三个并行的编码器 - 解码器流结构,类似于U - Net。将T1和T1ce、T2和FLAIR这两对相关的成像模态分别作为上、下两个流的输入,以更好地学习跨模态特征表示。中间流用于整合不同模态的互补知识。
- 自适应跨特征融合模块(ACF模块)
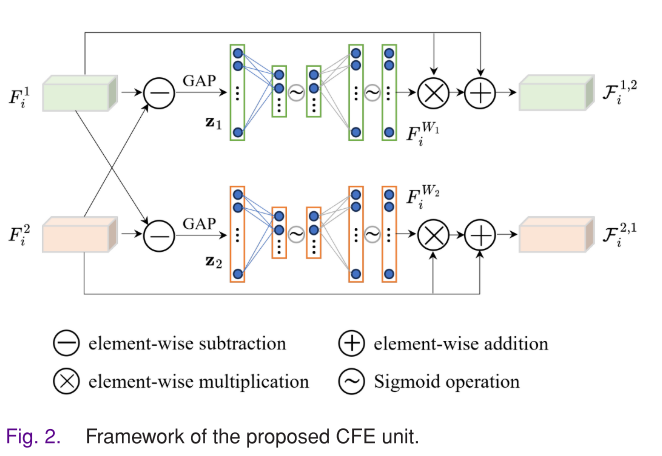
- 相关特征探索单元(CFE单元):该单元有两个并行分支,通过交叉引导策略探索不同模态特征表示之间的相关性。以一个分支为例,先计算两个模态特征的差值,经全局平均池化(GAP)得到通道统计信息,再通过两个全连接层挖掘通道依赖关系,最终得到包含通道权重的特征,用于突出不同模态之间的差异。另一个分支则交换输入特征的角色,以从不同角度突出差异。
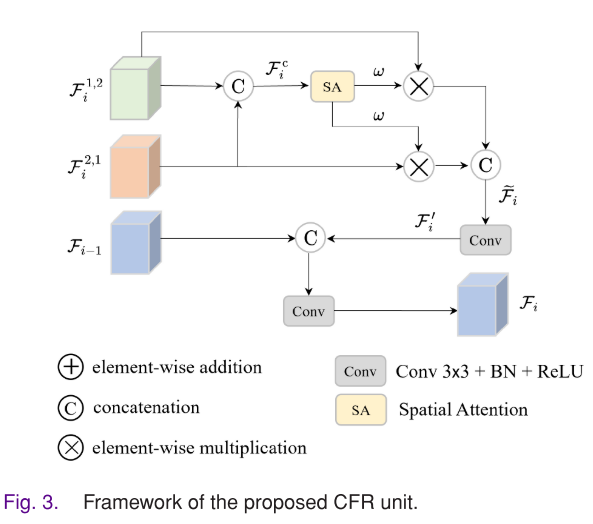
- 跨特征细化单元(CFR单元):将CFE单元两个分支的输出沿通道方向拼接,通过空间注意力(SA)块获得空间域的像素权重,对拼接特征进行加权,再经过拼接、卷积操作得到细化后的特征。同时,将该特征与相邻低层CFR单元的输出整合,得到最终输出。
- 预测不一致引导模块(PIG模块):利用上、下两个流预测结果的不一致性来引导网络关注易出错区域。在解码阶段,将中间流对应ACF模块的输出与预测不一致信息沿通道方向拼接,经过卷积等操作,使网络聚焦于易出错区域,减少不确定分割,最终生成准确的预测结果。
- 损失函数:ACFNet的总损失由三个流的损失组成,每个流的基本损失Lb是经典的Dice损失和交叉熵损失的线性组合。总损失公式为Lt = Lb(P1, G) + Lb(P2, G) + Lb(Po, G),其中G是每个场景的真实标签集,包含整个肿瘤、增强肿瘤和肿瘤核心的标签。 实验结果表明,ACFNet在多模态脑肿瘤分割任务中表现出色,优于六种主流的医学图像分割方法。消融实验也验证了ACF模块和PIG模块对整体性能提升的重要贡献。
实验(Compared with SOTA)
- 数据集:选用BraTS 2020数据集评估ACFNet。因该数据集测试部分不可用,仅使用训练部分,包含369个不同对象的多模态MR扫描,每个扫描有T1、T1ce、T2和FLAIR四种模态。从每个体积的轴向平面提取2D切片重建数据集,裁剪为224×224并归一化到[0, 1],按切片原始对象ID划分为80%训练集和20%测试集。
- 评估指标:采用四个广泛用于医学图像分割的评估指标,即Dice相似度系数、交并比(IoU)、灵敏度和第95百分位豪斯多夫距离(HD95)。
- 实现细节:在PyTorch框架上实现实验,在配备NVIDIA 3090 GPU和Intel 4210R CPU的Ubuntu服务器上训练和测试模型。使用SGD优化器,动量设为0.9,权重衰减设为1e - 4,初始学习率0.01并采用多项式衰减策略,批量大小12,训练100个epoch。
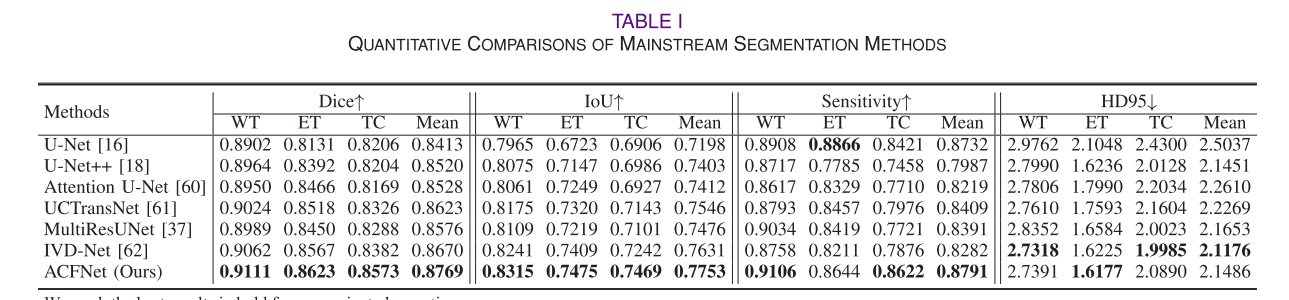
- 定量结果:将ACFNet与U - Net、U - Net++等六种主流医学图像分割方法比较。结果显示,ACFNet在多数评估指标上优于竞争对手。例如,在分割全肿瘤(WT)、增强肿瘤(ET)和肿瘤核心(TC)时,Dice值、IoU值和灵敏度值均有提升。单模态方法通常不如多模态方法,可能是直接级联所有模态会忽略其内在相关性,而在网络中间层融合多模态特征有助于发现和利用这些相关性以提高分割性能。此外,ACFNet计算效率方面的浮点运算(FLOPs)和推理时间高于多数竞争方法,但平行结构及ACF和PIG模块对捕捉模态差异、提高分割精度很关键。
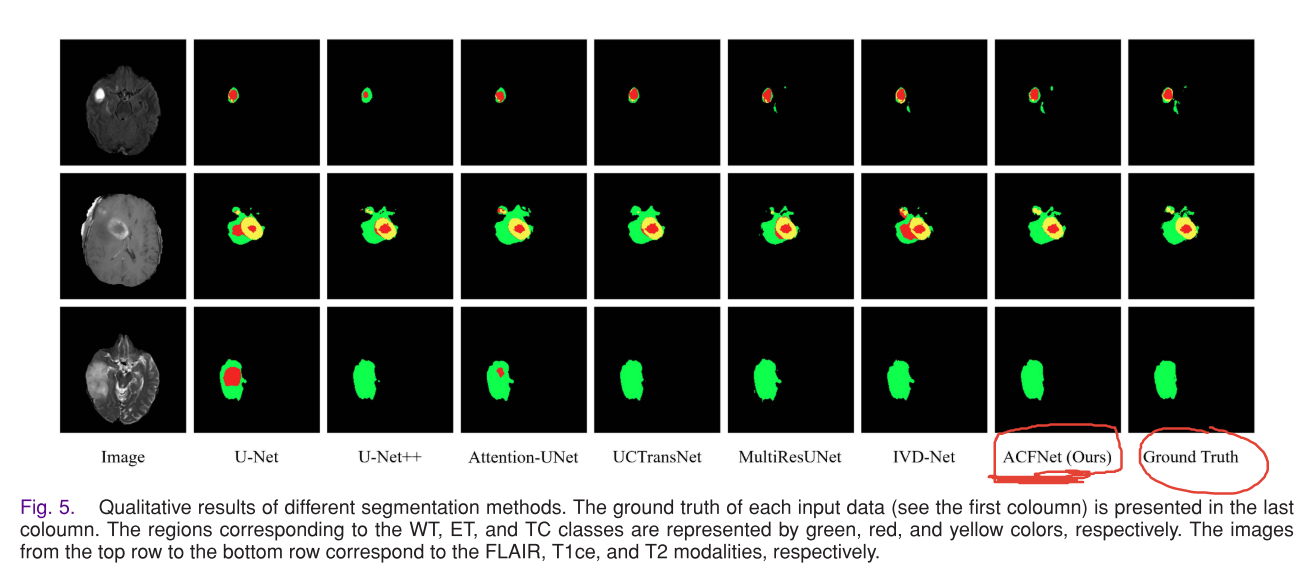
- 定性结果:通过可视化结果直观比较分割性能。单模态方法处理背景噪声能力有限,难以准确定位不同大小和形状的脑肿瘤,处理小病灶时易出现漏检。多模态方法因在多模态融合上的努力产生更准确结果,ACFNet在定位小病灶、区分病灶区域与周围环境方面比IVD - Net更准确,且在不同大小和形状的病灶上表现稳定,这得益于其有效的多模态特征融合和减少模态差距的策略。
实验(Ablation Experiments)
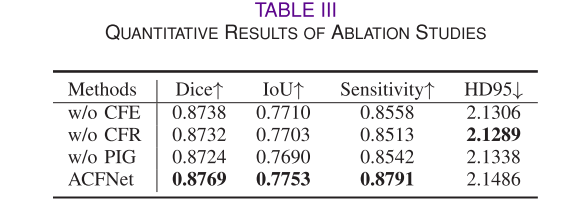
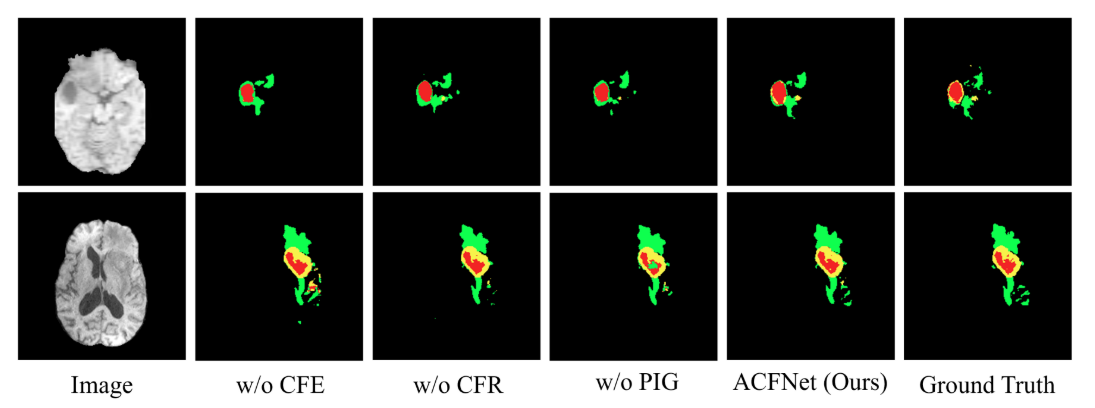
对ACFNet的两个关键模块ACF和PIG进行消融实验,每次仅移除考虑的模块,保持网络其余部分不变学习新模型。结果表明,ACF模块的CFE和CFR单元以及PIG模块对整体性能提升有积极贡献,可视化结果也显示这两个模块有助于获得准确的分割结果。
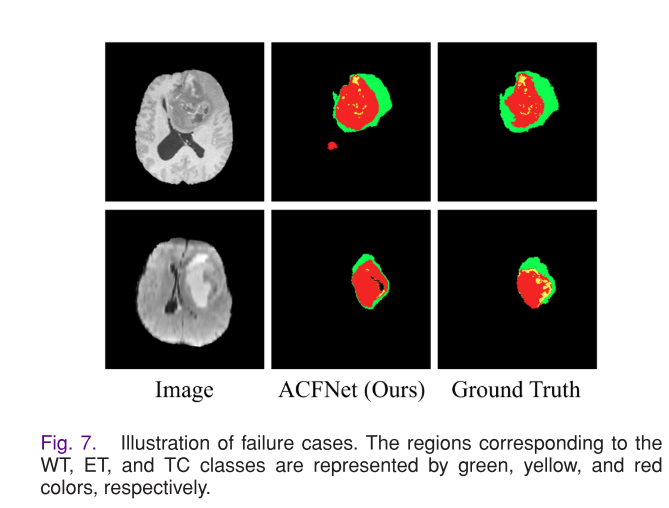
失败案例
ACFNet在一些具有挑战性的场景中无法准确分割脑肿瘤,如无法准确定位大肿瘤的精细细节,在肿瘤边界模糊时分割性能不佳。可能原因是ACFNet在这些场景中对复杂肿瘤的表示能力有限,特别是肿瘤与非肿瘤区域相似度高以及肿瘤区域内部差异大时。
结论
近年来,多模态数据中的自动脑肿瘤分割受到了越来越多的关注。本文介绍了一种名为ACFNet的新型深度神经网络,用于在磁共振成像(MRI)中实现准确的脑肿瘤分割,其动机主要体现在两个方面:
- 直接整合来自不同模态的中间特征可能会导致信息掩蔽,高效融合多模态数据有助于获得更具判别性的特征表示并提高分割性能。
- 正确发现多模态数据中的差异并利用这些差异来引导网络,有助于减少可能的假阴性或假阳性结果。 为了有效融合多模态数据,ACFNet使用了自适应交叉特征融合(ACF)模块来探索中间层的内在互补性;为了获得准确的分割性能,ACFNet利用了预测不一致性引导(PIG)模块,该模块能根据不同模态预测结果之间的不一致性,引导网络关注容易出错的区域。
实验结果表明,ACFNet可以有效解决多模态脑肿瘤分割问题,取得了令人满意的结果。此外,消融实验结果也验证了ACF模块和PIG模块的有效性。 基于本文的研究,未来的研究方向主要有两个:
- 探索ACFNet在除MRI之外的其他多模态图像分割任务中的性能。
- 开发降低ACFNet计算需求的方法,使其更有可能在临床环境中部署。



